SilencerDB沉默子元件数据的来源
首先从物种来看,主要包括人类和小鼠的数据资源。
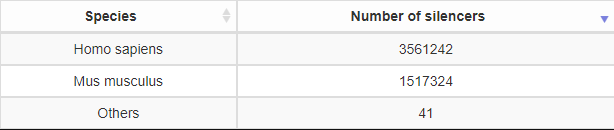
从组织器官覆盖度来看,Silencer数据也是覆盖比较全面的。
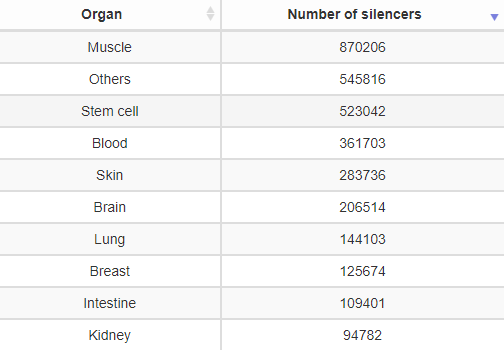
从数据来源的方法来看,可以看到绝大部分数据来源于计算方法预测,只有非常少的部分数据来源于高通量或低通量的实验验证。值得注意的是,其中实验验证的Silencer元件基本来源于Blood样本。
这篇文章里,我主要介绍SilencerDB数据库建立涉及到的计算方法的原理。
一、使用H3K27me3-DHS活性和基因表达的关联来预测Silencer
H3K27me3-DHS指与H3K27me3 peaks重叠的染色质开放区域,其活性和开放染色质的覆盖深度相关。该研究采用的是至少在三个细胞系中出现的H3K27me3-DHS peaks。该方法从两个方面来关联H3K27me3-DHS活性和基因表达:一是H3K27me3-DHS和邻近基因表达的相关性分析,二是H3K27me3-DHS和Hi-C远程作用的基因表达的相关性分析。
,代表该区域H3K27me3-DHS存在;代表该区域H3K27me3-DHS不存在。
表示该区域H3K27me3-DHS存在和不存在时,对应基因表达的差异显著性(Wilcoxon秩和检验P-value)
是一个示性函数,描述了该区域H3K27me3-DHS存在和不存在之间,对应平均基因表达水平的差异,示性为正相关,负相关或者不相关。
,该H3K27me3-DHS和对应基因表达显著负相关;
,该H3K27me3-DHS和对应基因表达显著正相关;
其余情况,该H3K27me3-DHS和对应基因表达无显著相关。
该研究也通过eQTL基因和转录抑制因子的富集,验证了correlation预测的silencer集合。
基于MPRA(大规模平行报告分析)验证关联分析预测的silencer集合
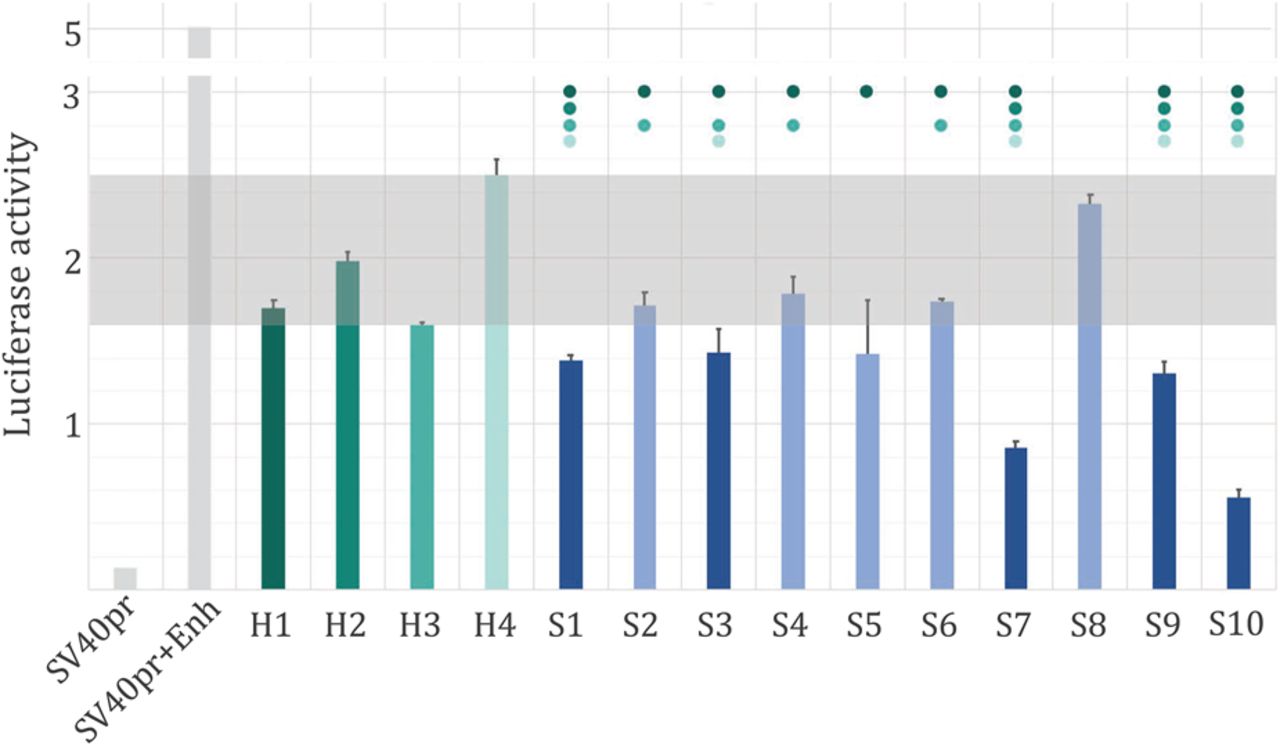
该实验选用了10个negCOR silencers(S1…S10)和4个基因表达无显著相关的H3K27me3-DHS(H1…H4),用K562细胞(an enhancer + SV40 promoter + luciferase reporter gene)作为基因报告实验体。结果如图所示,14条测试序列都表现出显著抑制基因表达的效果,即使是先前与基因表达无显著关联的H3K27me3-DHS区域。这个实验的不足还是实验过少,而预测的结果太多。
二、基于SVM的沉默效应分类器
上文H3K27me3-DHS活性和对应基因表达的关联性分析,确定了一些H3K27me3-DHS的调控属性,和基因表达正相关的类增强子元件,和基因表达负相关的类沉默子元件,以及无调控属性的区域。
正集:negtive correlation H3K27me3-DHS;负集:positive correlation H3K27me3-DHS;
H3K27me3-DHS用该区域的putative TFBSs,组蛋白修饰信号密度,邻近基因表达水平的最大值和最小值编码。
模型:linear kernel SVM;least squares; fivefold cross-validation.
应用以上基于negtive correlation H3K27me3-DHS和positive correlation H3K27me3-DHS构建的线性SVM模型,对所有训练样本的分类进行校正;并用构建好的模型对其余H3K27me3-DHS区域进行分类评分,划定阈值,和negtive correlation H3K27me3-DHS相似的区域划分为Silencer。控制FDR <= 0.1。
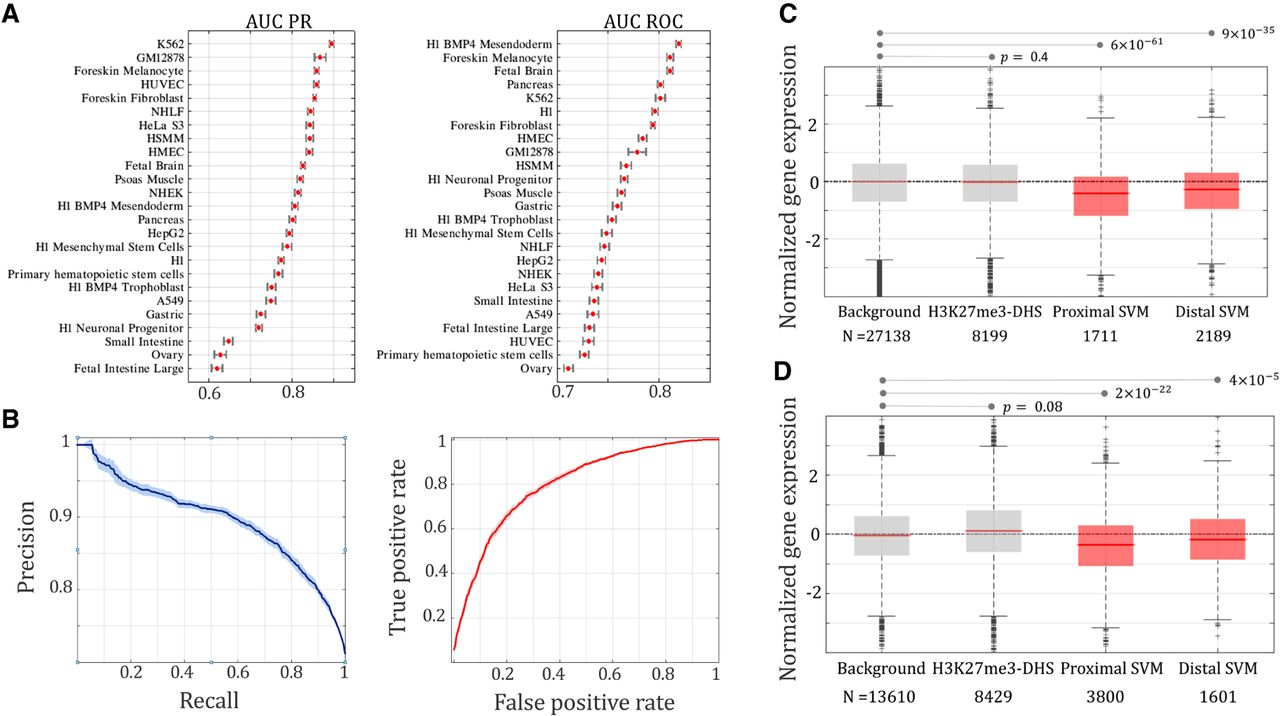
图A为不同组织类型中silencer预测AUC值,范围0.62到0.89;ROC值范围0.71到0.82;图B为K562细胞中的AUC和ROC曲线。
该研究根据Hi-C连接情况,将SVM silencer关联的基因分为两组:“distal SVM gene”,这类基因仅有一个SVM silencer存在Hi-C连接,并且该SVM silencer被一个及以上的基因给分隔开;另一类为“proximal SVM gene”,这类基因由至少一个SVM silencer通过Hi-C产生连接,并且这些SVM silencer位于基因内部(内含子区)。个人觉得这里很奇怪,关联的基因应该还有其它类型,比如多个远端silencer Hi-C连接的gene。
因为之前构建SVM模型的时候,采用了邻近基因的表达水平作为特征,所以这里可以用Hi-C连接的远端基因的表达水平来验证SVM预测的silencer。如图C所示,通过Hi-C连接的“proximal SVM gene”和“distal SVM gene”的基因表达水平都显著低于背景水平和H3K27me3-DHS整体水平。约有65%的SVM silencer相关的远端基因的表达水平显著低于随机期望(65% of distal SVM genes versus 50% of all genes, binomial test P < 10−100)。同样,针对eQTL关联的基因去验证,可以发现SVM silencer关联基因的显著低水平表达(图D)。
同样,针对SVM预测的silencer,研究也通过转录抑制因子和除H3K27me3以外的其它抑制型组蛋白修饰信号的富集,来验证。
三、基于gkm-SVM的沉默效应分类器
- 采用simple subtractive analysis方法定义uncharacterized CREs
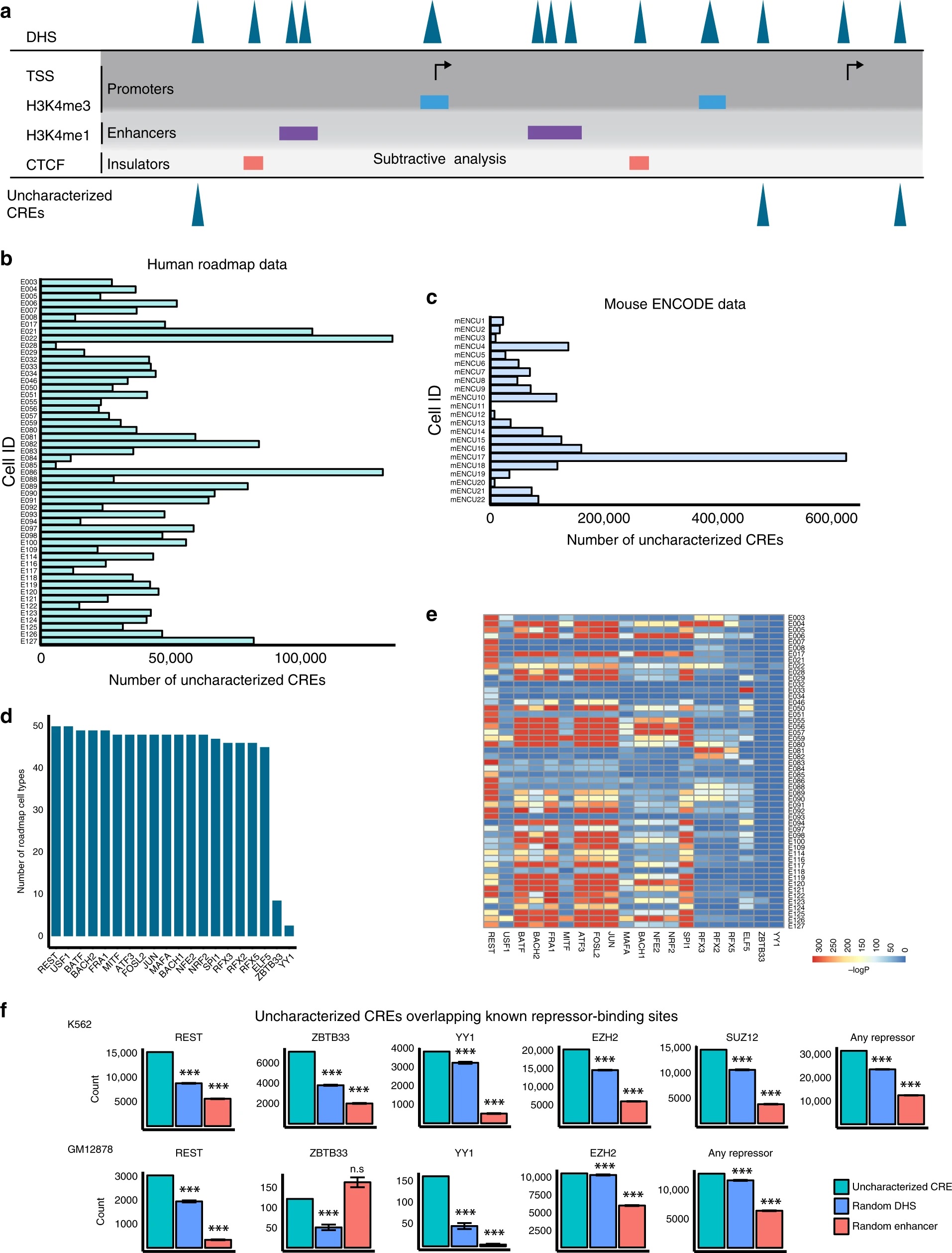
将不同细胞类型基因组DHS染色质开放区,除去已知promoter(2.5 kb window around TSS and H3K4me3 peaks),enhancer(H3K4me1 peaks)或者insulator CTCFBS(CTCF sites)区域,剩下的这部分未知区域,定义为uncharacterized CREs。该研究从Roadmap和ENCODE数据库中总共获得了82个人类细胞类型中2,315,105 uncharacterized CREs,22个小鼠细胞类型中1,299,866 uncharacterized CREs,用于silencer元件的鉴定和预测。 - 对uncharacterized CREs进行抑制型转录因子motif和抑制型转录因子结合位点富集分析,发现该区域存在显著富集
- MPRA实验验证部分uncharacterized CREs
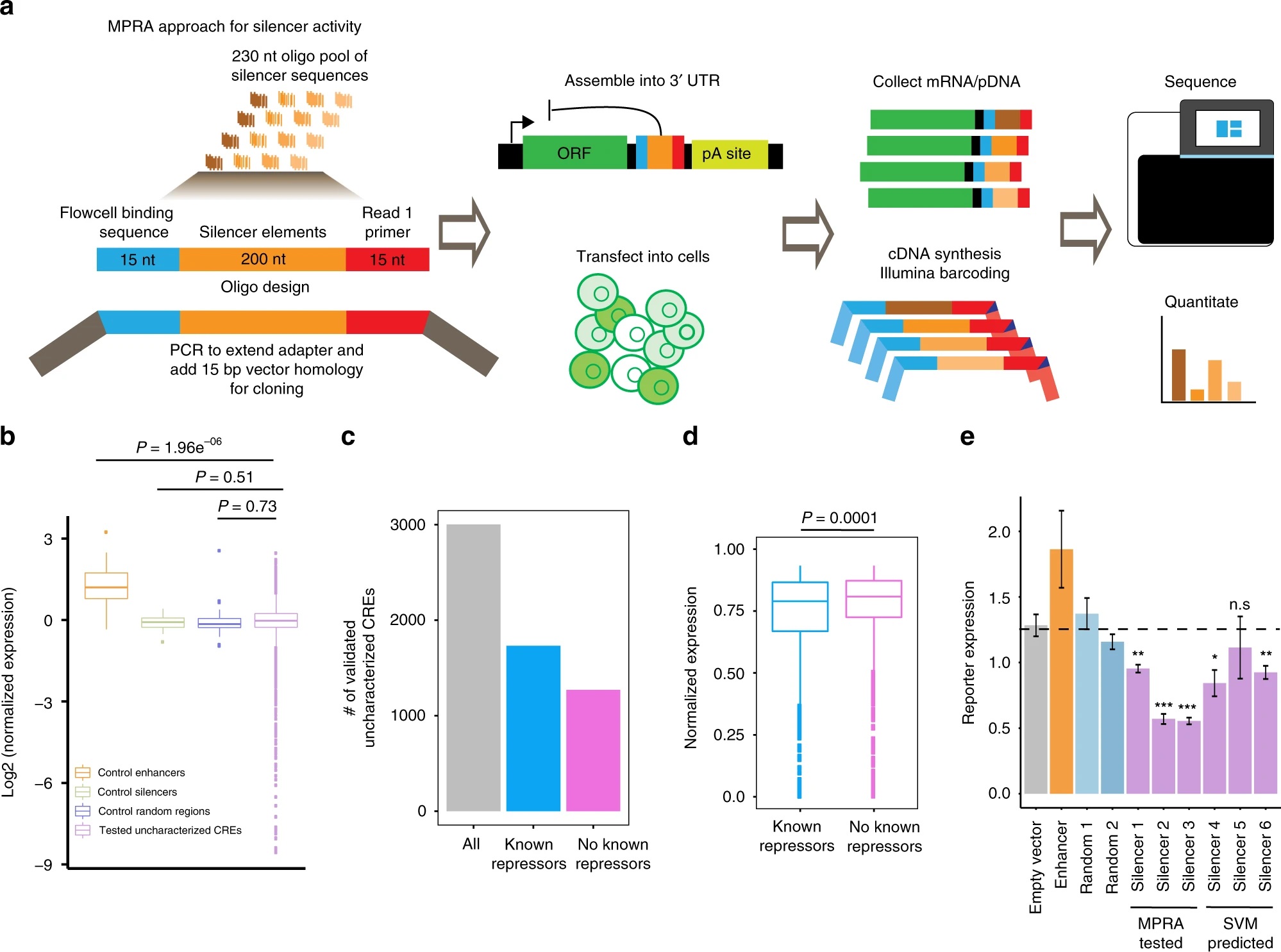
实验设计:k562细胞; 基于STARR-seq的MPRA;super core promoter (SCP1);7430 uncharacterized CREs(其中3705个至少含有一个已知抑制型TFBS,另外3725个至少含有一个抑制型转录因子motif), 20 known silencer elements, 20 known enhancer elements, and 67 randomly selected regions(control sets)
总共在7430 uncharacterized CREs找到活性水平低于随机控制组的uncharacterized CREs3001个,定义为沉默子(silencer )。
随后该研究也从传统的基因报告实验(SCP1+luciferase gene;K562细胞),和CRISPR-Cas9基因编辑实验(敲除uncharacterized CREs,评估对基因表达的影响)对潜在silencer进行了验证,结果发现这些uncharacterized CREs基本属于沉默元件。 - 潜在沉默子元件预测
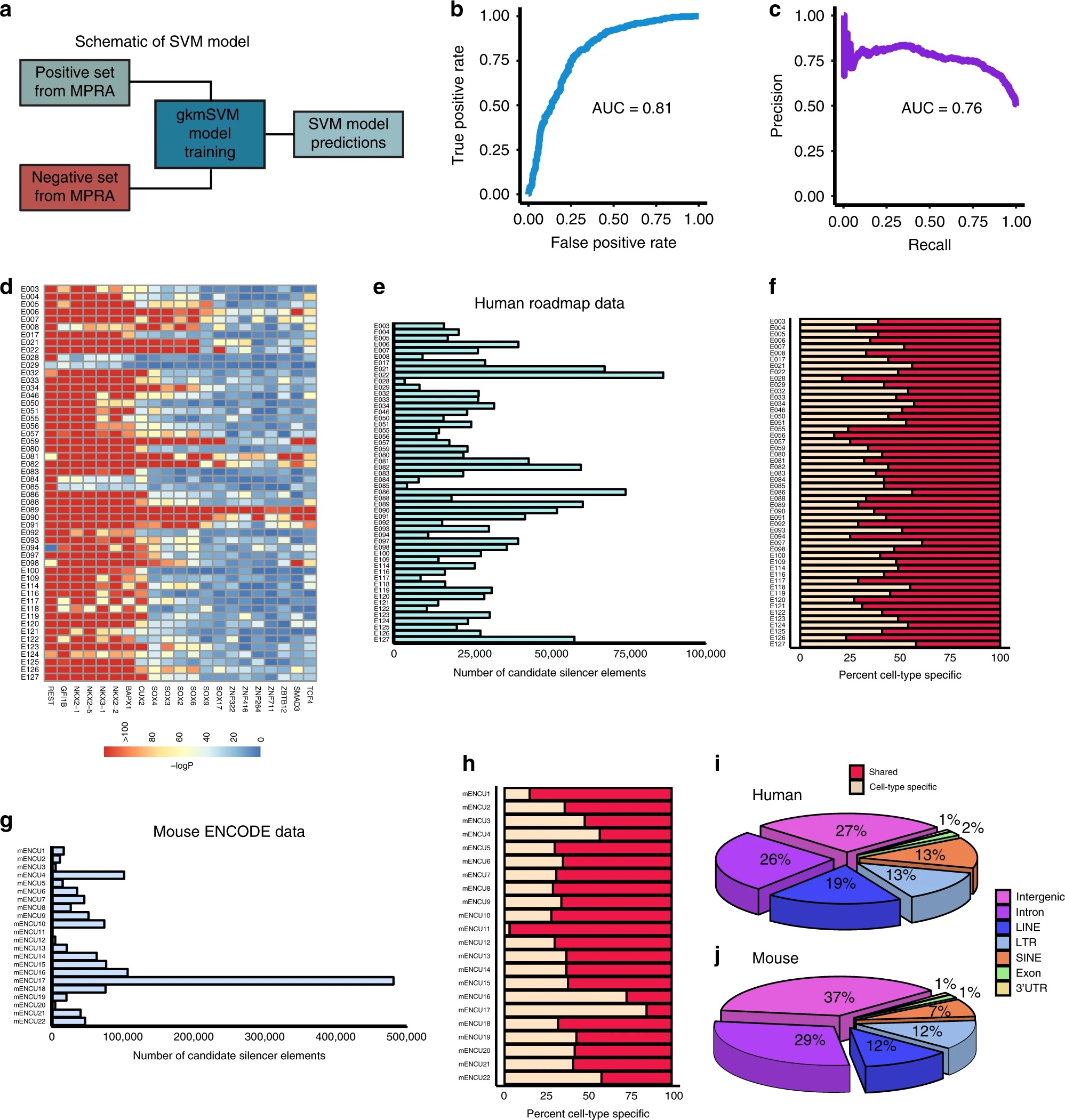
正集:the top 2000 uncharacterized CREs sequences with the lowest MPRA activity (K562 cells)
负集:the bottom 2000 uncharacterized CREs sequences with highest MPRA activity (K562 cells)
模型:gkm-SVM:80%(trainning),20%(test)
选择准确度最高的模型用于silencer预测。
该研究也提取了对数据有区分度的特征集,发现这部分特征集富集抑制型转录因子motif。
该研究在人类82个细胞组织类型中总共预测了1,706,989 candidate silencer elements,在小鼠22个细胞组织类型中预测了965,198 candidate silencer elements。
四、基于深度学习模型的框架
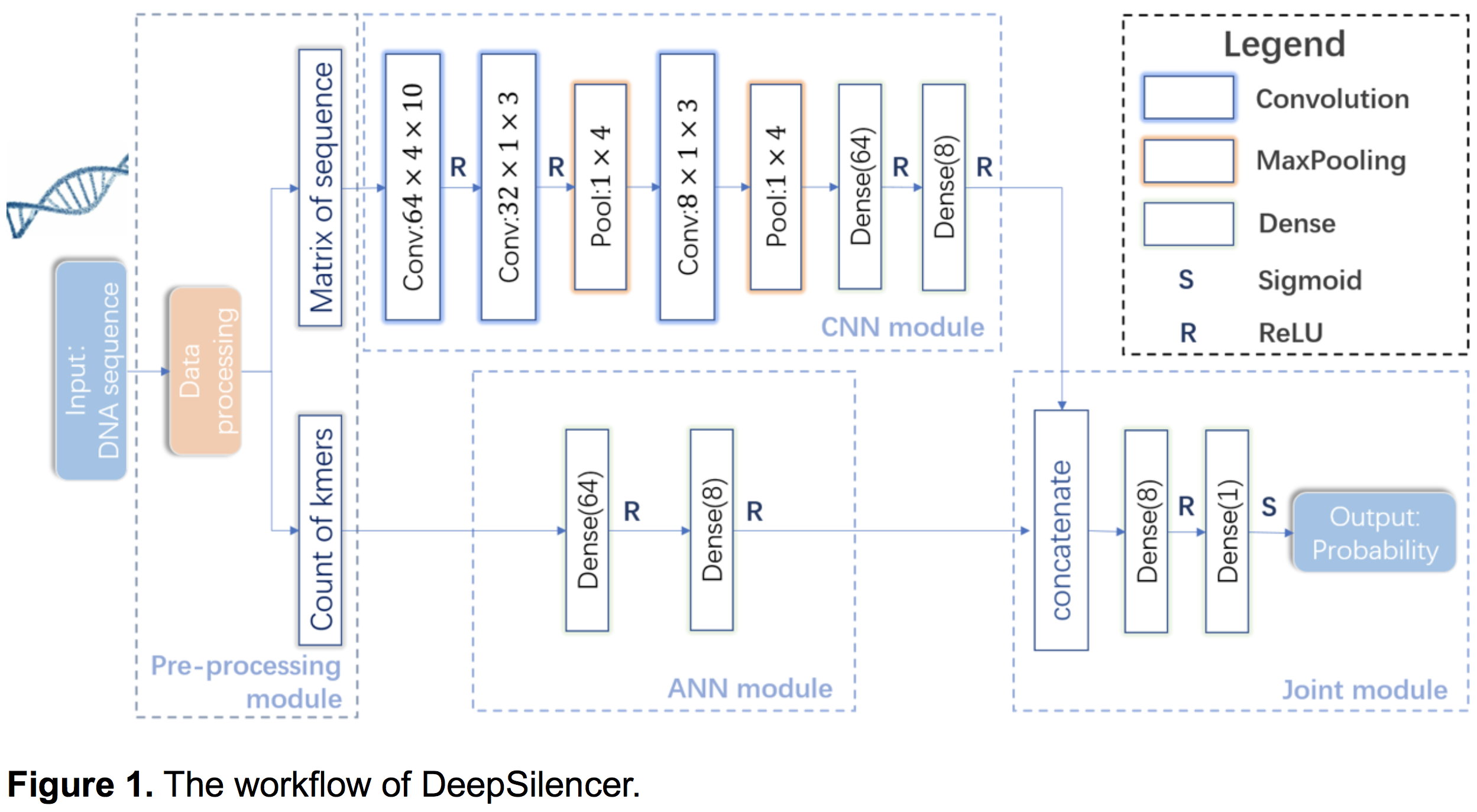
注意事项:
基于SVM silencer预测的方法使用的训练数据集来源于correlation分析的结果,因此correlation分析会比SVM预测的结果更可靠,correlation分析的准确性也会影响SVM预测的准确性。
References:
- http://health.tsinghua.edu.cn/silencerdb/download.php
- Huang D, Petrykowska HM, Miller BF, Elnitski L, Ovcharenko I. Identification of human silencers by correlating cross-tissue epigenetic profiles and gene expression. Genome Res. 2019;29(4):657-667.
- Doni Jayavelu, N., Jajodia, A., Mishra, A. et al. Candidate silencer elements for the human and mouse genomes. Nat Commun 11, 1061 (2020).