EPDnew 数据库promoter的来源

启动子是特定基因转录的DNA区域,启动子一般位于基因的转录起始位点,5'端上游,启动子长约100-1000bp。 在转录过程中,RNA聚合酶与转录因子可以识别并特异性结合到启动子特有的DNA序列(一般为保守序列),从而启动转录。这里主要讲人类启动子数据。该数据库包括两部分:一是蛋白编码基因的启动子序列——Hs_EPDnew_006;二是非编码基因的启动子序列——HsNC_EPDnew_001。
一、Hs_EPDnew_006蛋白编码基因启动子数据库的构建
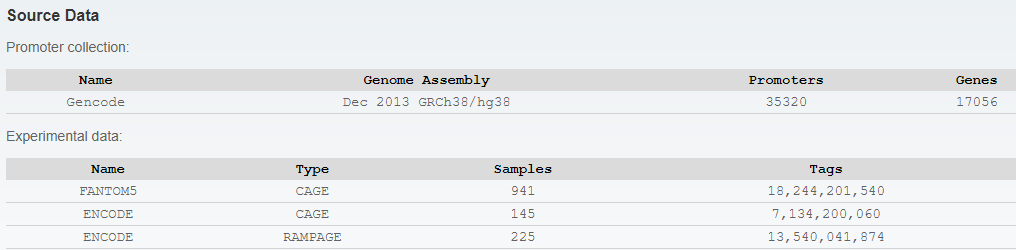
- 获取GENCODE注释的启动子数据
根据'gene_type' = 'protein_coding'确定蛋白编码基因的编码本位置信息。采取严格质控策略,保证每个转录本(包括剪切位点连接的转录本)至少有一个确定的mRNA数据支持。具有相同TSS位置的转录本数据进行合并,编码到一个转录本下。总共得到覆盖17056个蛋白基因的35320 个TSS records信息。 - GENCODE TSS转录起始位点信息收集
上一步从GENCODE收集的TSS转录起始位点信息整理格式如下:- NCBI/RefSeq chromosome id
- "ENSEMBL"
- position
- strand ("+" or "-")
- "1"
- TranscriptID..GeneName
- CAGE-seq数据的整合
从UCSC和FANTOM5数据库获取CAGE-seq数据(鉴定TSS和定量RNA表达),将hg19 alignment转换为hg38 alignment后,转为bed基因组位置信息。 - CAGE peak calling
对每个CAGE和RAMPAGE数据,按照以下参数进行peak calling:- Window width = 1
- Vicinity range = 200
- Peak refine = N
- Count cutoff = 9999999
- Threshold = 5
- RAMPAGE(TSS mapping)数据分析
ENCODE bam -> peak calling -> peak selection -> quality control - Gencode+RAMPAGE TSS validation dy CAGE data
Gencode+RAMPAGE TSS设定为CAGE peak富集的200bp window,或者定位到基因5'-UTR区域并且有至少5个tags。然后移动TSS到高密度tag区域进行校正。如果表达水平低于更强基因特异性起始位点的 10% 或低于 10 个标签,则丢弃具有多个 TSS 的基因的二级启动子。 - 不同样本promoter数据的整合
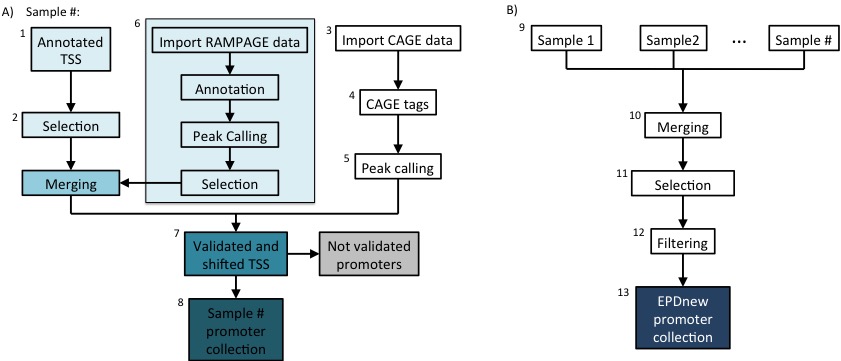
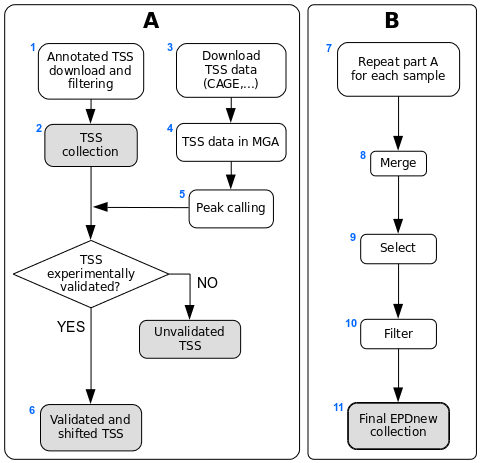
不同样本数据可能产生不同的启动子集合,对于同一个转录本也可能对应不同的起始位点,因此进行整合校正并且去除冗余非常重要。思路见图B。 - 样本特异的启动子集合的质控
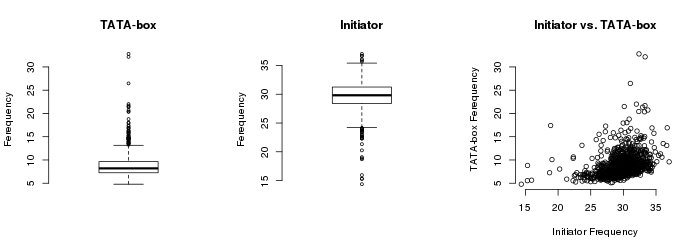
在进行最终收录前,需要对每个样本的启动子集合的质量进行质控,排除低质量样本。为了实现这一点,每个启动子集合都根据TATA-box和Inr基序在预期位置(各自处于TSS 的 -29bp的位置和在TSS区域)的分布进行评分。丢弃具有非常低基序频率(Inr 频率 < 10% 和 TATA-box < 5%)的样品(总共 3 个样品)。下图显示了 TATA-box 和 Inr 在所有特定样本启动子集合中的分布:
- 整合启动子集合并进一步TSS选择
整合到一个文件中的不同样本的启动子集合,仅保留至少有三个样本支持的转录本启动子。
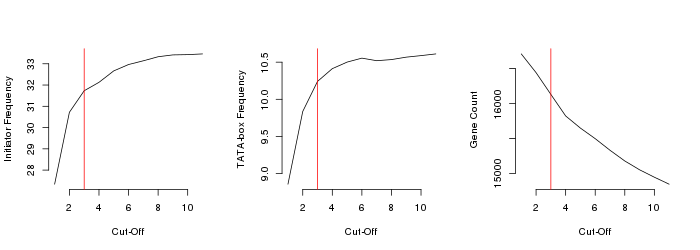
由多个样本验证的转录本可能会将 TSS 设置在更广泛的区域而不是单个位置。为了避免这种不一致,对于每个转录本,我们选择了被大量样本验证的位置作为真正的 TSS。
mapping到同一基因(100 bp 窗口)的其他 TSS 附近的转录起始位点被合并到一个独特的启动子中,遵循相同的规则:保留由更多样本验证的启动子。 - EPDnew启动子集合统计
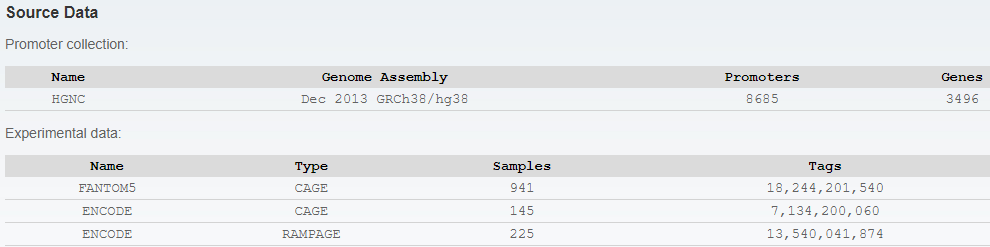
总共29598 个实验验证的启动子数据被收录。
二、HsNC_EPDnew_001非编码基因启动子数据库的构建
References:
- https://epd.epfl.ch//index.php
- https://epd.epfl.ch/epdnew/documents/HsNC_epdnew_001_pipeline.php
- https://epd.epfl.ch/epdnew/documents/Hs_epdnew_006_pipeline.php