ChromHMM 功能基因组注释基本原理
根据先验的表观遗传学研究结论,不同染色质印记标记下的DNA序列具有不同的功能特征。H3K4me3和活性启动子相关,维持启动子CpG低甲基化;H3K36me3和基因本体相关,维持基因本体高甲基化;H3K9me3和异染色质区域高甲基化维持相关;H3K27me3介导的基因沉默和DNA甲基化是竞争性抑制作用,相互替换通过会维持基因沉默,但有时DNA甲基化替换PRC介导的H3K27me3会激活基因表达。H3K27me3基因沉默比DNA甲基化可塑性更强(Greenberg et al., 2019)。H3K4me1和启动子和增强子有关(Bae et al.,2020),可方便粘连蛋白的募集,从而促进增强子和启动子长程相互作用(Yan et al., 2018);H3K27ac和活性增强子相关(Creyghton et al., 2010)。活性调控元件往往出现在开放染色质区域。
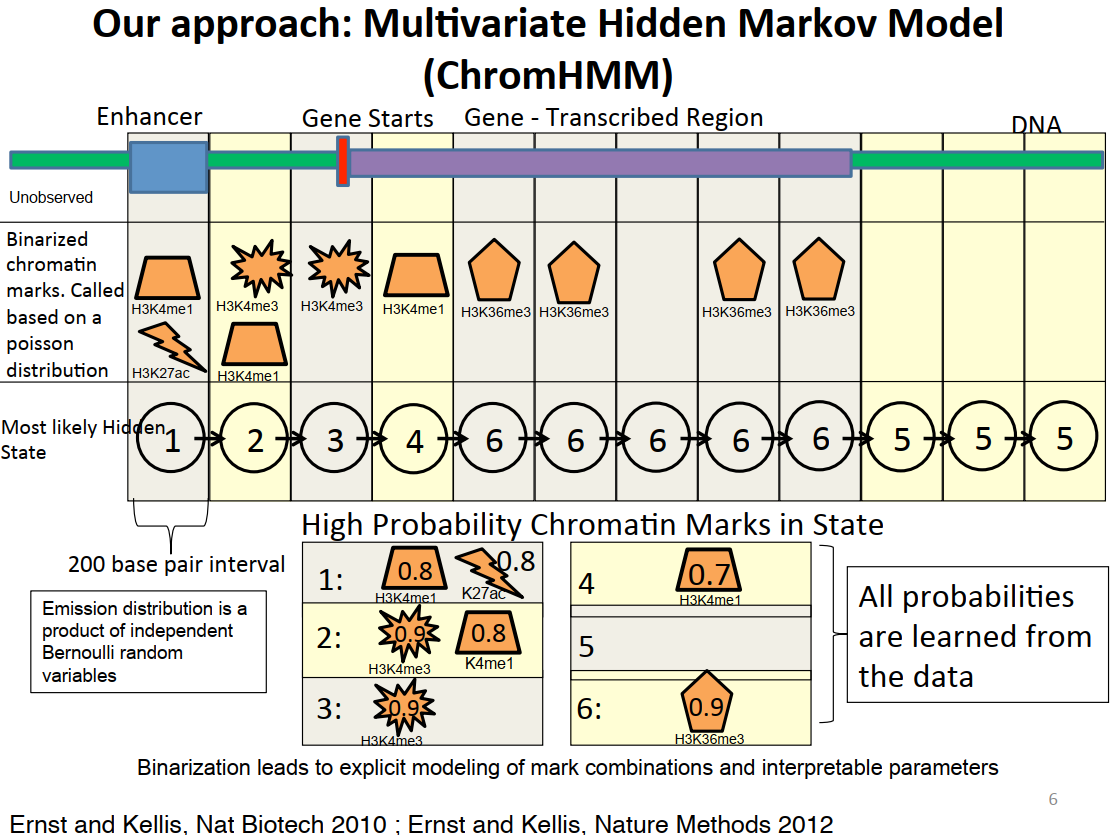
在这种背景下,Ernst和Kellis等人采用多变量隐马尔可夫生成模型,以200bp为单位,计算基因组范围内每个状态单元内可能性最高的状态,再结合先验背景知识对染色质状态进行注释,为启动子、增强子,转录本区域等等。可以设置状态数目,调节注释精度,如ENCODE计划利用9种染色质Marks对9个人类细胞系功能基因组进行注释,采用15-states模型,15个状态为9种Marks中的一种或几种的组合;又如Ernst and Kellis et al., 2015 整合Roadmap和ENCODE共127个细胞组织类型,分别利用5个染色质Marks(15-states model)、6个染色质Marks(18-states model),12个染色质Marks(25-states model)对染色质状态进行了注释,其中25-states模型注释的是ChromImpute推断的染色质状态数据。

至于方法里为什么默认采用200bp为状态单元,一方面是因为200bp是差不多一个核小体单位,另一方面200bp接近ChIP-seq reads长度的中位数。这样的状态单元一定程度上能反映组蛋白修饰的作用范围和转录因子的作用范围。当然作者也提到用户也可以根据自己需求重新定义状态单元长度。
参考文献:
1)Greenberg, M.V.C., Bourc’his, D. The diverse roles of DNA methylation in mammalian development and disease. Nat Rev Mol Cell Biol 20, 590–607 (2019).
2) Bae S, Lesch BJ. H3K4me1 Distribution Predicts Transcription State and Poising at Promoters. Front Cell Dev Biol. 2020;8:289. Published 2020 May 5. doi:10.3389/fcell.2020.00289
3) Yan J, Chen SA, Local A, et al. Histone H3 lysine 4 monomethylation modulates long-range chromatin interactions at enhancers. Cell Res. 2018;28(3):387. doi:10.1038/cr.2018.18
4) Creyghton MP, Cheng AW, Welstead GG, et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proc Natl Acad Sci U S A. 2010;107(50):21931-21936. doi:10.1073/pnas.1016071107