连锁不平衡
连锁不平衡在群体遗传学中,指同一条染色体上两个等位基因在传代中共同遗传的概率。连锁不平衡程度往往会受到同源染色体间重组率的影响。重组率越高,连锁不平衡程度越小。
连锁不平衡在概率统计上,也反映为同一条染色体上两个等位基因的非随机相关性。因此,假设等位基因A,B,则它们之间的连锁关系可以表示为群体中观测AB基因型频率和随机背景下(Hardy-Weinberg平衡)AB基因型频率之差。
DAB=pAB−pApB
具体来说,
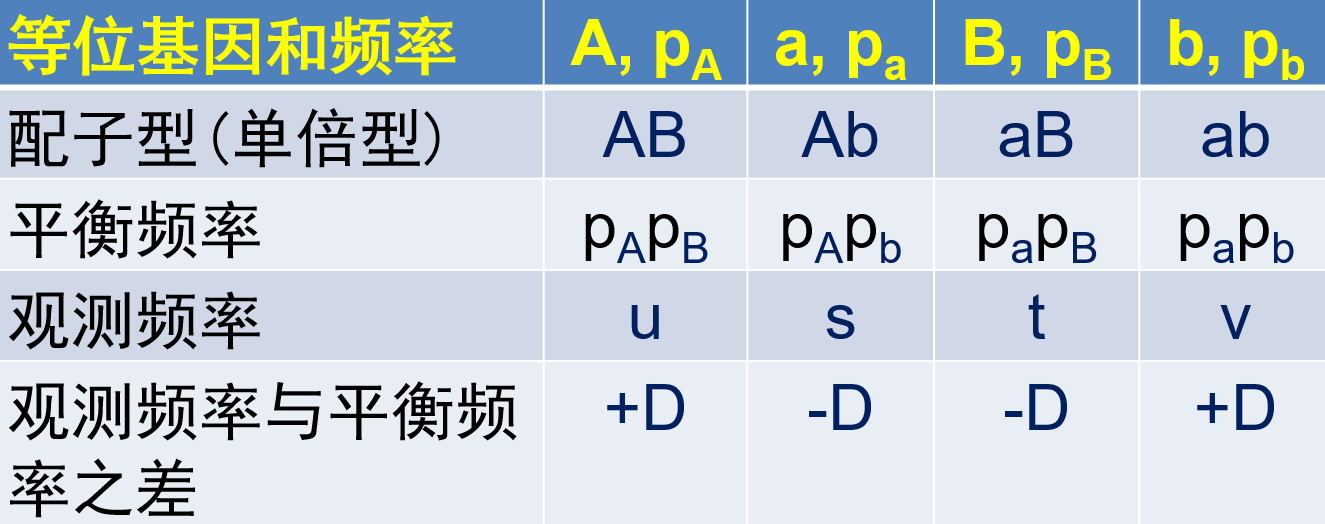
基因频率: pA=u+s; pa=t+v; pB=u+t; pb=s+v
对于AB来说, D=pAB−pApB=u−(u+s)(u+t)=u(1−u−s−t)−st=uv−st
连锁不平衡的其它度量方法(1)
当uv−st>0时:
Dmax=min[pApb,papB]
当uv−st<0时:
Dmax=min[pApB,papb]
标准化不平衡度Dˊ定义为:
D′=DmaxD
连锁不平衡的其它度量方法(2)
将不平衡度平方后除以四种等位基因频率的乘积:
r2=pApapBpbD2
- r2不平衡度的平方根正好等于两个座位间的相关系数;
假设biallelic site A: 两个等位基因的频率分别为 pA,pa,满足pA+pa=1
biallelic site B: 两个等位基因的频率分别为 pB,pb,满足pB+pb=1r=Var(pA)×Var(pB)Cov(pA,pB)
在样本大小为n的群体中,随机抽取等位基因使其满足频率为pA,则pA的分布为2n次随机抽样的二项分布,pA的方差为2npApa 。pB的分布同理。两者的协方差为2n(pAB−pApB)r=(2n×pA×pa)×(2n×pB×pb)2n×(pAB−pA×pB)
由D的定义可知,DAB=pAB−pApB
则上式可写成:
r2=4n2×pA×pa×pB×pb4n2×DAB2=pApapBpbDAB2
- r2不平衡度乘以总样本量正好等于不平衡检验的 χ2统计量。
因此,不平衡度参数r2在自然群体的遗传分析中有更广泛的应用。
总结:
- 前面的三种不平衡度参数,各自从不同侧面反映座位间的不平衡程度,难以相互取代。
- 后两者参数的目的是消除基因频率对不平衡度的影响。但是,不平衡度和基因频率都是从配子观测频率计算出来的,这度量方法有时也难以完全排除等位基因频率的影响。
- 此外,这些参数都不能用来判断不平衡度的显著性,不平衡度的显著程度仍然要借助于统计假设检验。
References:
https://www.isbreeding.net/teaching/?type=detail&id=14
