一款基于permutation tests的基因组区域关联分析计算软件
关于permutation tests的介绍,可以见https://socrates1100.github.io/post/permutation-test/
本文主要侧重介绍它的基本函数功能。
- permTest
pt <- permTest(A=my.regions, B=target.regions, randomize.function=randomizeRegions,
evaluate.function=numOverlaps)
A是参考基因组区域;B指感兴趣的研究区域。这个函数会计算B intervals在A中的富集程度,即B与A overlap的intervals数目相较于随机背景,是否具有显著高或低的差异。
该函数采用常用来检验指定的基因组区域是否具有更高的甲基化水平
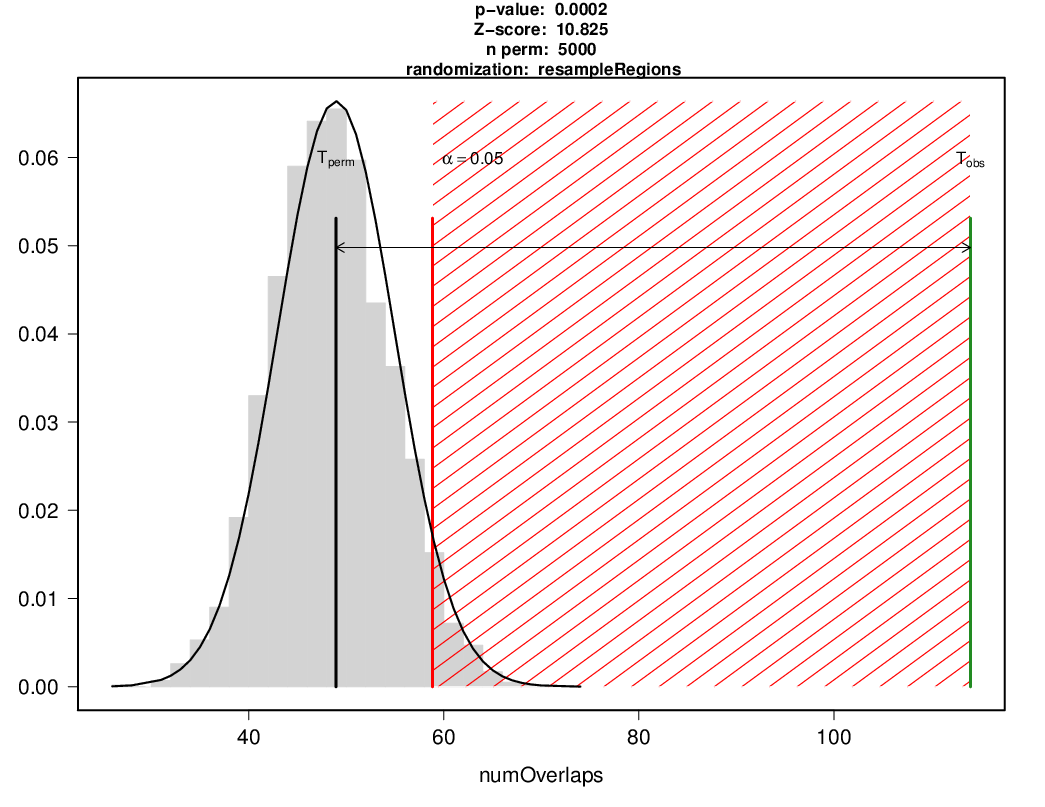
- overlapPermTest
set.seed(123)
overlapPermTest(A, B, ntimes=10, mc.set.seed=FALSE, force.parallel=TRUE)
为了保证代码多线程运行时,结果的可重复性。需要在函数开始前设置一次随机数,在函数内部关闭随机数生成。这个函数相当于permTest函数采用了
- localZScore
lz <- localZScore(A=A, B=B, pt=pt, window=10*mean(width(A)), step=mean(width(A))/2 )
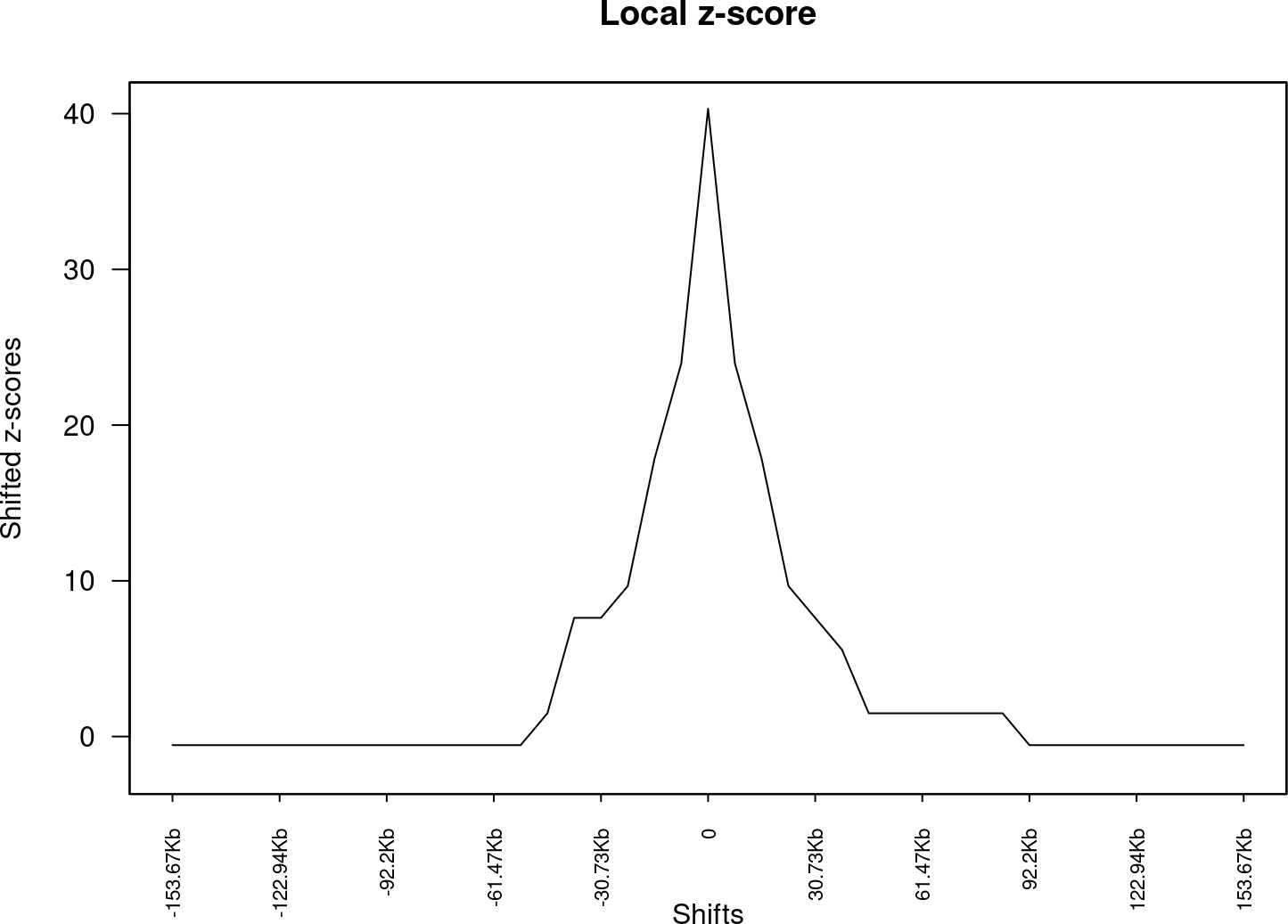
References:
https://bioconductor.org/packages/release/bioc/vignettes/regioneR/inst/doc/regioneR.html#how-to-perform-a-permutation-test-with-regioner